生长素在植物的生长和发育中起着至关重要的作用。植物可以快速感知并响应生长素水平的变化,这些应答反应主要涉及2类生长素应答基因,即生长素/吲哚-3-乙酸(Aux/IAA)基因家族和生长素应答因子(auxin response factor,ARF)基因家族[1]。研究表明,Aux/IAA家族蛋白可以与ARF形成多个二聚体,在植物发育中具有多种作用,例如根系发育、枝条生长和果实成熟[2]。AUX/IAA家族的成员已被鉴定为短寿命核蛋白,包含4个高度保守的结构域(结构域Ⅰ、Ⅱ、Ⅲ和Ⅳ)[3]。AUX/IAA家族蛋白具有与ARF相互作用或抑制或激活ARF基因转录的特定结构域。在低生长素浓度条件下,Aux/IAA蛋白与ARF因子结合并阻止生长素应答基因的激活。在高生长素水平下,Aux/IAA蛋白被泛素化,随后通过与生长素信号传递受体F-BOX(TIR1/AFB)相互作用的26S蛋白酶体结合而降解[4]。被释放的ARF因子可调节下游生长素应答基因的表达。通常,不同的TIR1/AFB与AUX/IAA蛋白组合具有不同的生长素结合亲和力,并且生长素水平在不同的组织和发育阶段有所不同,从而导致不同的生长素感应作用[5]。生长素水平的时空动态变化可以转化为基因重编信号,精确地调节植物生长和发育的过程。OsIAA10基因的功能缺失突变体在经过30 d的暗光诱导培养后,无法形成正常愈伤组织,且下胚轴不受生长素抑制而伸长[6]。但是其下游作用于哪个ARF因子,ARF因子下游作用的具体基因,尚不清楚。
CRL1启动子区域包含2个推定的生长素反应元件(AuxRE)。AuxRE基序特异性作用于水稻ARF,并充当CRL1表达的顺式基序[7]。CRL1基因编码1个侧根形成正调节因子,其表达受生长素信号传导途径中的ARF直接调节,但具体的ARF因子还未知。在CRL1的根起始功能模型中,生长素调控AUX/IAA的降解,并且AUX/IAA与ARF相互作用,释放的ARF与CRL1启动子中的AuxRE相互作用,触发侧根起始区域的CRL1基因转录,从而导致根形成起始[8]。本研究通过基因敲除、超表达结合诱导、酵母杂交等研究方法,确定生长素通路中的相互作用因子,揭示水稻愈伤组织形成与其根发育途径的关联性,以期为进一步研究愈伤发生机制打下基础。
1 材料与方法
1.1 试验材料
采用粳稻品种中花11进行遗传转化,并以中花11黄化幼苗用于原生质体分离。载体构建过程中用到的菌株与载体参见文献[6]。
rTaq酶、dNTP等常用PCR试剂与限制性内切酶购于TaKaRa公司,组培试剂乙酰丁香酮、潮霉素、水解酪蛋白和2,4-D 等购自Sigma公司,引物合成和测序等工作均由生工生物工程(上海)公司 承担,详见文献[6]。
1.2 CRL1基因敲除载体的构建
选择CRL1基因序列上 CDS 区的2个靶位点,分别为pYLCRISPR/Cas9-MH 多靶点载体构建体系的U3靶点和 U6a 靶点,构建gRNA表达盒,核酸序列为GGCCACCATGAACAACTGGCTGG,ACGACGGCGACGATGGCTGGTGG(核酸序列5′-3′) 。
1.3 CRL1基因超表达载体的构建
以pCAMBIA1300s(35S启动子)超表达载体作为克隆载体的骨架,在载体的多克隆位点处插入CRL1基因的基因组全长序列。PCR扩增出的目的片段使用试剂盒纯化回收,以BamHⅠ、KpnⅠ双酶切载体及目的片段,回收酶切产物[1]。之后再通过NEB T4-连接酶的连接体系将目的片段连接至线性化载体。得到的连接产物转化DH5a感受态细胞,培养过夜后挑取生长健康的单克隆菌落至对应抗性的3 mL LB液体培养基中,过夜培养,抽出质粒后测序鉴定[9]。
1.4 农杆菌侵染法转化水稻
将测序无误的终载体电转化到农杆菌中,通过挑取单克隆抽提质粒,测序鉴定阳性菌落,筛选培养过程参见文献[6]。
1.5 转化植株分子检测
取CRL1基因敲除并过表达转化植物的叶片,CTAB法提取DNA [9]。
1)对转基因植物进行阳性检测。以提取的DNA样品为模板,引物为潮霉素通用引物, PCR程序参见文献[6]。
2)植物基因组编辑检测。在2个目标点外侧200~400 bp处设计引物,通过扩增出来的条带的大小判断基因敲除是否成功[6]。
1.6 CRL1纯合敲除和超表达T1种子的愈伤组织诱导
选择成熟饱满一致的CRL1基因纯合敲除和过表达的T1种子,以野生型作为对照。在正常的诱导培养基中,除去颖壳并挑选状况良好且无病原体感染的种子。 将40粒种子浸泡于75%乙醇30 s,然后用无菌蒸馏水洗涤[2]。 加入0.15%HgCl2溶液,浸泡15~30 min,最后用无菌蒸馏水冲洗,直到白色泡沫消失。每瓶培养基中接种8粒种子,每个处理接种5瓶;在黑暗中于28 ℃培育30 d[6]。
1.7 OsIAA10的亚细胞定位
取生长15 d左右的水稻幼苗制备原生质体细胞,并构建OsIAA10-GFP融合表达载体。参照文献[9],于激光共聚焦显微镜下观察融合蛋白在原生质体中的表达部位。
1.8 OsIAA10互作蛋白筛选
构建OsIAA10基因的诱饵载体,并与空文库载体PPR3-N共转化酵母菌株NMY51,在SD/-Trp/-Leu/-His/-Ade培养基中培养,进行自激活检测[1],膜蛋白文库筛选过程参照文献[6]进行。
1.9 酵母杂交点对点互作验证实验
构建的重组质粒AD-OsARF和BD-OsIAA10共转化至酵母AH109中,涂布于SD/-Trp/-Leu平板上[1]。菌落生长后,选择少量单个菌落,并将其溶解在一定量的灭菌ddH2O中。混合后,用移液器吸取3 μL的菌液点于SD/-Trp/-Leu和SD/-Trp/-Leu/-His/-Ade筛板(3个生物学重复样本)。在30 ℃恒温培养箱中培养3 d后,观察酵母的生长。如果白色菌落在SD/-Trp/-Leu和SD/-Trp/-Leu/-His/-Ade平板上生长,则表明OsARF和OsIAA10蛋白存在相互作用。如果白色菌落仅能在SD/-Trp/-Leu平板上生长,而不能在另外的平板上生长,则表明OsARF与OsIAA10蛋白没有相互作用[1]。
2 结果与分析
2.1 CRL1敲除和超表达载体的构建与检测
CRISPR终载体质粒构建(图1A)成功后,阳性质粒串联的gRNA表达盒用MluⅠ酶切(图1B),验证无误后再测序确认。将扩增的CRL1基因全长序列插入到超表达载体(图1C),以KpnⅠ、BamHⅠ酶切检测。
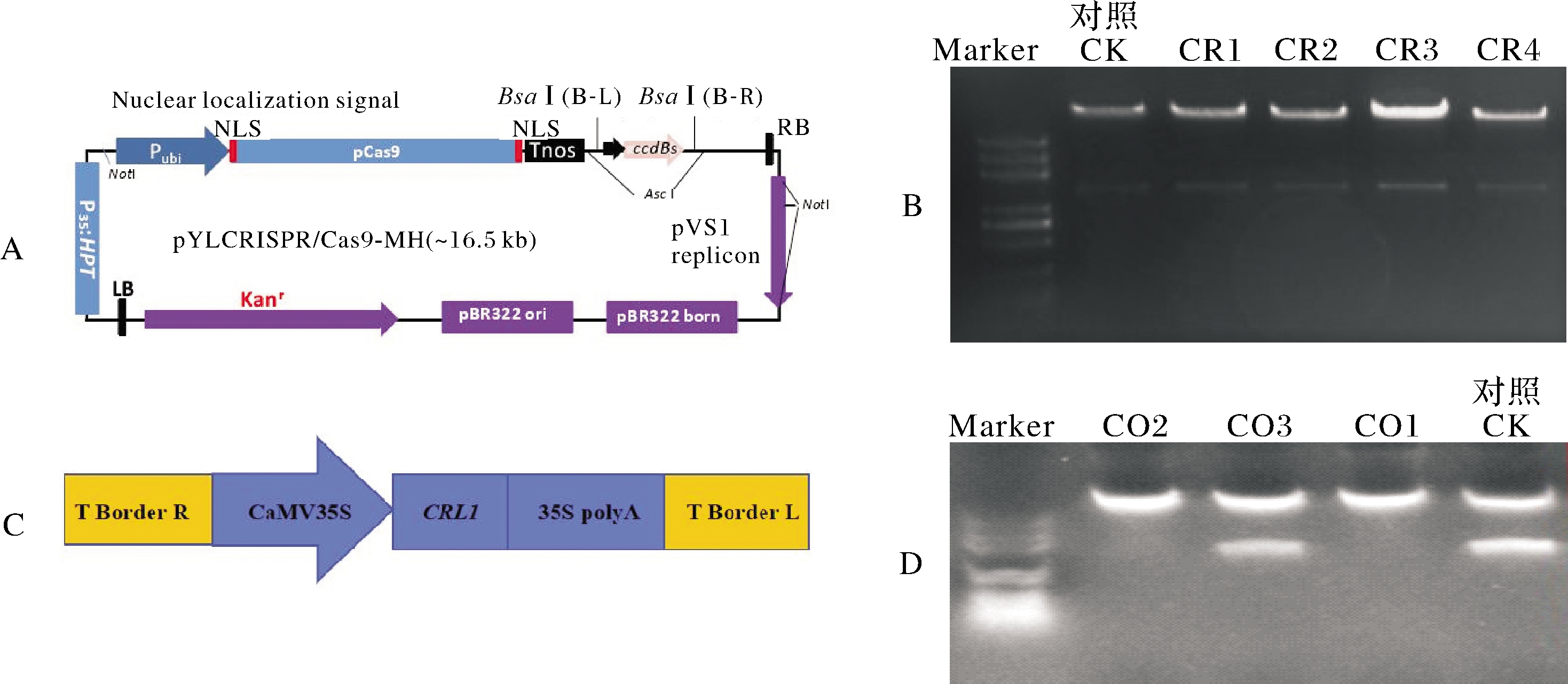
A.CRISPR 载体构建; B.MluⅠ酶酶切CRISPR 终载体质粒的琼脂糖凝胶电泳; C.超表达终载体示意图; D.琼脂糖凝胶电泳检测表达终载体。A. CRISPR vector diagram; B. Agarose gel electrophoresis image of CRISPR final vector plasmid digested by MluⅠ; C.Schematic diagram of overexpression final vector; D. Agarose gel electrophoresis image of overexpression final vector restriction detection.
图1 载体示意图以及酶切检测结果
Fig.1 Vector schematic diagram and enzyme digestion test results
2.2 敲除和超表达载体的遗传转化
将构建的CRL1基因的CRISPR/Cas9 载体和超表达载体转入农杆菌菌株 EHA105,转化中花11愈伤,经过2轮潮霉素筛选,得到的转化苗进行hpt阳性检测,CRL1基因敲除转化苗进行基因编辑检测。CRL1基因的基因敲除突变体 T0代得到8个大片段缺失突变家系,超表达突变体T1代得到113株阳性突变苗(图2)。
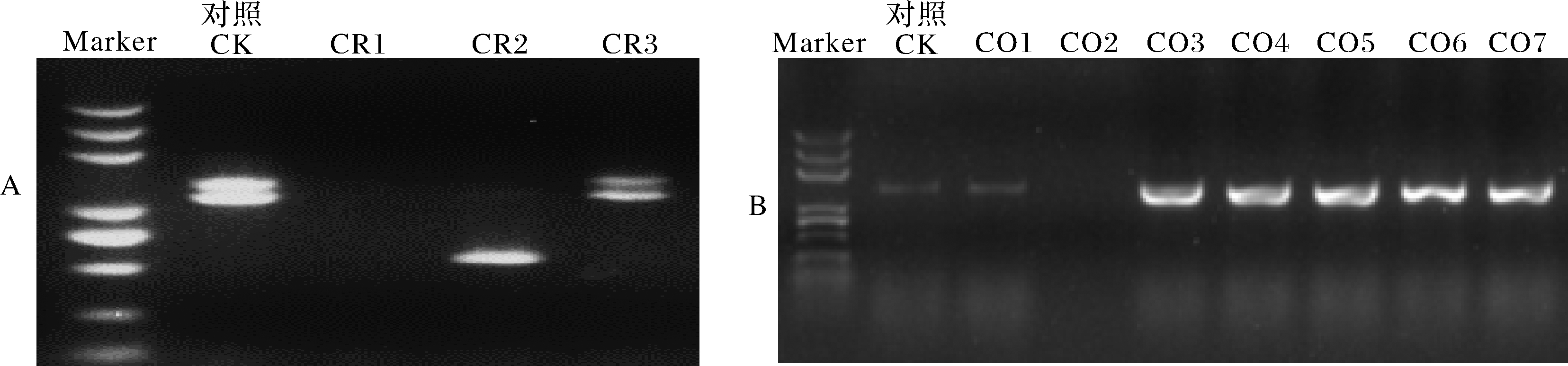
A.基因敲除突变体编辑结果的PCR检测结果;B.超表达突变体PCR检测结果。A.PCR detection results of the editing results of gene knockout mutants;B.PCR positive detection results of overexpression mutants.
图2 基因敲除和超表达的分子检测的琼脂糖凝胶电泳
Fig.2 Agarose gel electrophoresis image for molecular detection of gene knockout and overexpression
2.3 CRL1纯合敲除和超表达水稻种子的愈伤诱导
愈伤诱导结果显示,CRL1的纯合超表达家系种子并无显著的诱导率与诱导速率变化;CRL1基因敲除家系种子表现出明显不同表型,在诱导15 d后CRL1突变体种子未出现不规则的细胞团,只有下胚轴的伸长。在诱导30 d后,对照组已产生健康的愈伤组织,而CRL1敲除突变体的种子没有完整的愈伤组织(图3)。暗示着CRL1基因可能是水稻种子愈伤诱导中的关键因子。
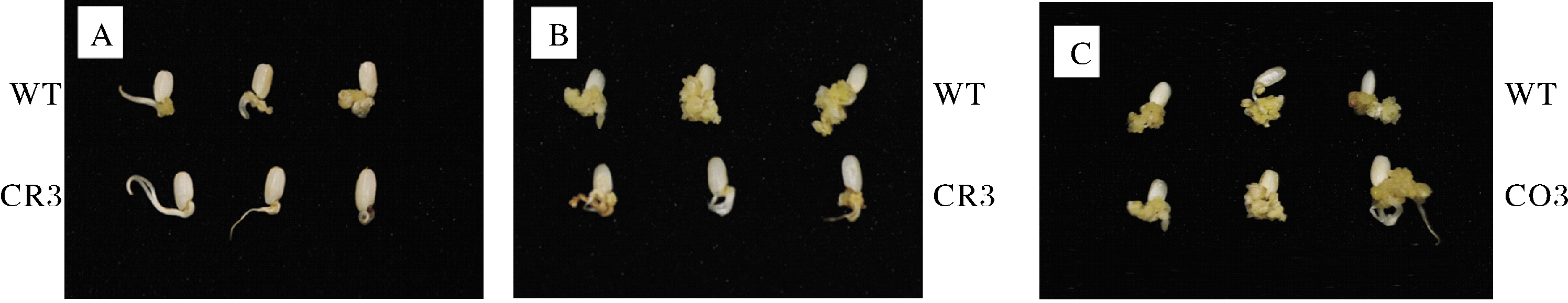
A:诱导15 d后的野生型种子与敲除突变体种子; B:诱导30 d后的野生型种子和敲除突变体种子;C:诱导30 d后的野生型种子和超表达突变体种子。 A:Wild-type seeds and knockout mutant seeds 15 days after induction; B:Wild-type seeds and knockout mutant seeds 30 days after induction;C:Wild-type seeds and overexpression mutant seeds 30 days after induction.
图3 野生型水稻种子与水稻CRL1基因敲除突变体CR3、超表达突变体CO3种子诱导表型对比
Fig.3 Comparison of seed induced phenotype of wild type rice and rice CRL1 geneknockout mutant CR3 and gene overexpress mutant CO3 seed induced phenotype
2.4 OsIAA10的亚细胞定位
将融合表达载体转化中花11原生质体,在培养12~16 h后,置于激光共聚焦显微镜下观察,结果显示OsIAA10蛋白定位于细胞核与细胞膜上(图4)。
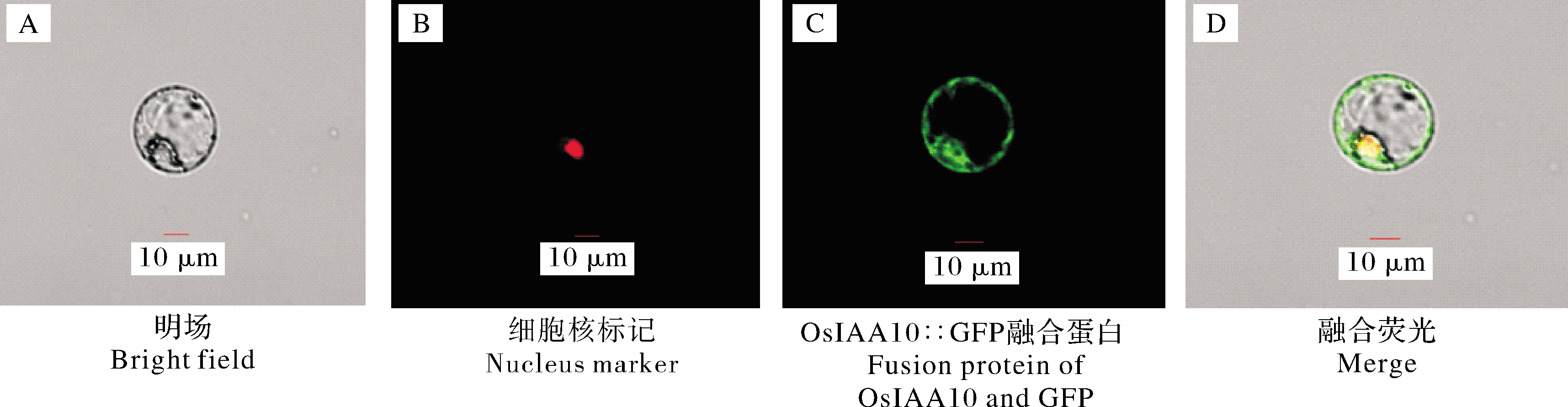
图4 OsIAA10在水稻原生质体中的亚细胞定位
Fig.4 Subcellular localization of OsIAA10 in rice protoplasts
2.5 OsIAA10互作蛋白筛选
DUAL膜系统文库中使用的cDNA是野生型中花11在整个生长期的各种器官和组织的cDNA[1]。由于不清楚蛋白的N端、C端定位情况,直接对3个载体进行自激活检测,结果显示OsIAA10只在pBT3-SUC载体中没有自激活现象(图5)。 对从相互作用阳性克隆中提取的质粒进行测序,并与水稻基因组数据库进行比较,以排除假阳性序列,获得了4个主要的候选相互作用蛋白(表1),都是OsARF家族的成员。
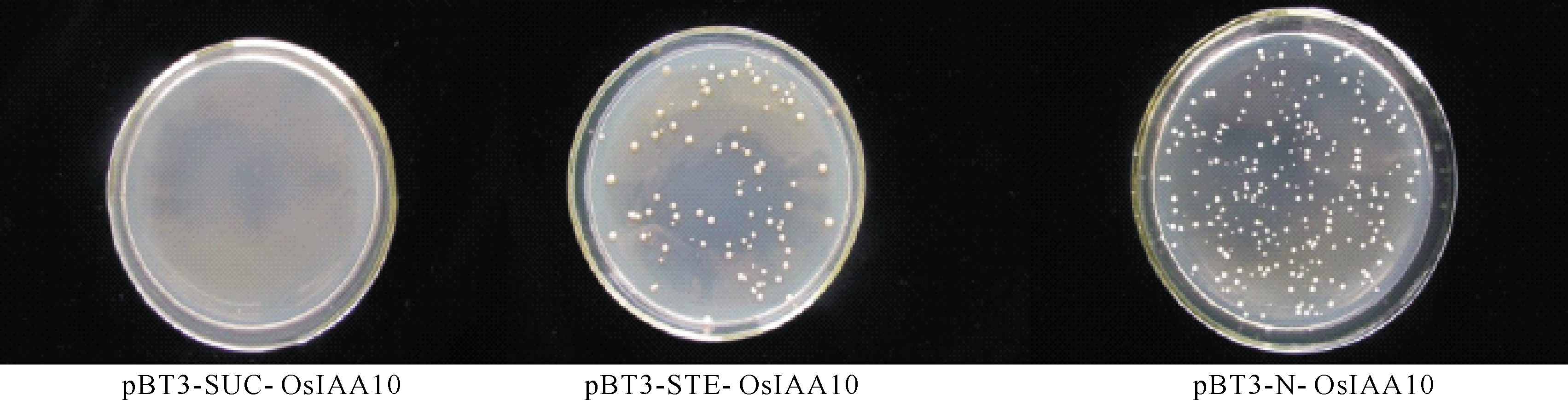
图5 pGBKT7-OsIAA10自激活检测
Fig.5 pGBKT7 - OsIAA10 self activated test
表1 筛库的候选互作蛋白信息
Table 1 Screening library candidate interaction protein information
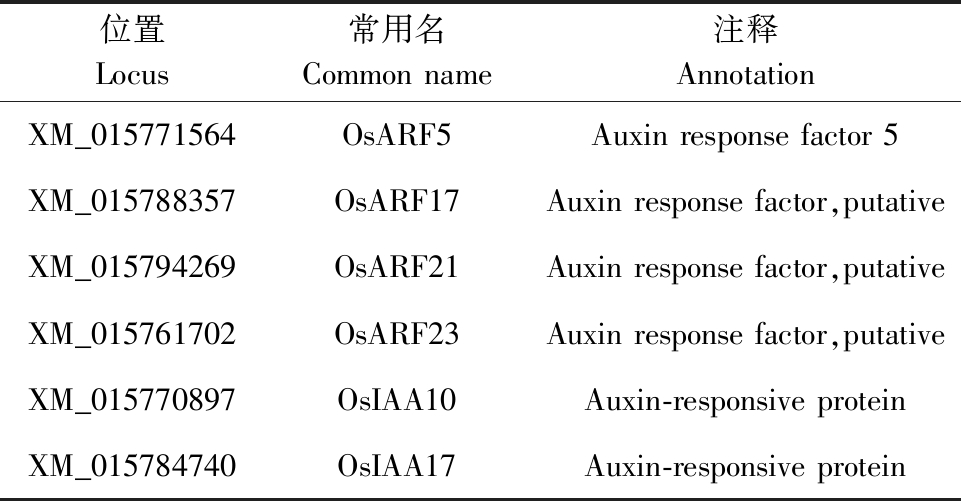
位置Locus常用名Common name注释AnnotationXM_015771564OsARF5Auxin response factor 5XM_015788357OsARF17Auxin response factor,putativeXM_015794269OsARF21Auxin response factor,putativeXM_015761702OsARF23Auxin response factor,putativeXM_015770897OsIAA10Auxin-responsive proteinXM_015784740OsIAA17Auxin-responsive protein
2.6 候选互作蛋白的验证
将OsARF5、OsARF17、OsARF21和OsARF23基因全长序列分别插入文库载体pPR3-N,构成4个筛选载体,与诱饵载体共转至酵母AH109菌株中,于SD/-Ade/-His/-Ura/-Trp平板培养。结果显示,OsARF5、OsARF17、OsARF21和OsARF23在酵母点对点验证实验中均能够和OsIAA10发生互作。

分别稀释10倍与100倍,第1行至第5行分别为:阳性对照、阴性对照、BD-IAA10加AD空载、 AD-ARF5加BD空载、BD-IAA10与AD-ARF5。Diluting 10 times and 100 times,the first row to the fifth row of the figure are:positive control,negative control,BD-IAA10 plus AD no-load,AD-ARF5 plus BD no-load,BD-IAA10 and AD-ARF5.
图6 酵母点对点实验
Fig.6 Some yeast point-to-point experiments
2.7 CRL1与候选基因点对点验证
采用酵母ABA杂交系统,将构建的含有转录因子基因的pGADT质粒分别转化到诱饵菌株和突变诱饵菌株,pGAD-p53质粒转化为p53-ABAi菌株作为阳性对照[1]。 将转化的酵母菌株涂在SD/-Leu培养基上,ABA的自激活抑制质量浓度为500 ng/mL, 结果显示,在没有ABA的培养基上,包含所有转化组合的酵母细胞可以正常生长。 在含有500 ng/mL ABA的培养基上,含有突变诱饵的酵母菌株根本无法生长,只有含有CRL1-ABAi+pGADT-OsARF5或CRL1-ABAi+pGADT-OsARF21或阳性对照组合的酵母细胞才可以生长。 基于此结果可以推断,转录因子OsARF5和OsARF21可以结合酵母中CRL1基因的启动子区域的基序并激活抗性基因的表达。

图7 酵母单克隆点对点实验(阳性克隆验证)
Fig.7 Yeast single-clone point-to-point experiments(proof of positive clone)
3 讨 论
本研究通过基因敲除、超表达突变体结合突变体种子诱导实验,证明了CRL1在调控水稻愈伤组织的形成中发挥重要作用,通过酵母双杂交筛库实验,筛选并验证了OsIAA10和OsARF5、OsARF17、OsARF21、OsARF23存在相互作用关系。通过酵母单杂交点对点实验,验证了其中的OsARF5、OsARF21能和CRL1启动子区的特定元件结合,以此调控CRL1基因的转录。初步证明了CRL1基因是生长素通路中OsIAA10基因下游的一个关键调控基因,初步探明了一条在水稻愈伤组织诱导初期发挥重要作用的生长素调控通路。CRL1基因下游对生理生化过程的影响机制仍然未知,但是单从表型足以认识到它的重要性。同时本研究中对CRL1基因的研究将生长素响应的愈伤组织诱导途径与冠根原基发育起始途径两者直接联系了起来[10]。这与同家族的LBD16、LBD18等同源基因在拟南芥中发挥的作用类似。同时超表达突变体种子诱导实验未发现异常,说明可能存在着未知的反馈调节通路[10-13]。
有研究显示水稻CRL1基因下游调控基因超过600个 [14-15],许多基因和参与侧根形成的拟南芥基因同源,但是其中大约有四分之一是水稻所特有的[16-18]。600多个基因中被上调表达的主要是涉及甲基化、转录调控、ROS和脂质代谢以及细胞壁水解与合成等反应过程的相关基因,下调的主要是与光合作用等反应相关的基因[19-21],这与愈伤诱导过程中的相关生化反应比较吻合。
进一步的研究可以从以下几个方面进行:(1)本研究结果显示CRL1与OsIAA10、OsARF5、OsARF21之间存在调控关系,而IAA家族与ARF家族之间存在着保守的互作模式,CRL1上游是否还存在与OsIAA17等因子的调控关系?我们将继续对此进行深入的研究。(2)不同的水稻品种对组织培养的耐受能力不同[22-23],CRL1在其中发挥的作用是否有差别,结合不同品种与激素条件,改善现有的优良水稻品种组培力,培育出优良的可组培品种,进一步研究籼稻与粳稻品种之间组培力差异的原因,促进优良籼稻品种的生物技术改造和利用[24-26]。(3)CRL1在愈伤诱导过程中的表达模式是否与根发育过程存在不同?虽然愈伤诱导早期与根系发育的早期原基形成都受到CRL1控制[27],但之后的生理发育过程却相去甚远,是下游的哪一个调控步骤造成如此差异?之后的研究可以从组织分生模式、细胞增殖、激素稳态、转录调控和细胞壁水解与合成[28-30]等方面开展。
[1] ANZOLA J M,SIEBERER T,ORTBAUER M. Putative Arabidopsis transcriptional adaptor protein (PROPORZ1) is required to modulate histone acetylation in response to auxin[J].PNAS,2010,107:10308-10313.
[2] ATTA R,LAURENS L,BOUCHERONDUBUISSON E. Pluripotency of Arabidopsis xylem pericycle underlies shoot regeneration from root and hypocotyl explants grown in vitro[J]. Plant journal,2009,57:626-644.
[3] YANG C,BRATZEL F,HOHMANN N,et al. VAL-and AtBMI1-mediated H2Aub initiate the switch from embryonic to postgerminative growth in Arabidopsis[J].Curr Biol,2013,23:1324-1329.
[4] WANG Q,XIE W,XING H,et al. Genetic architecture of natural variation in rice chlorophyll content revealed by a genome-wide association study[J]. Molecular plant,2015,8:946-957.
[5] BERCKMANS B,VASSILEVA V,SCHMID S P C. Auxin-dependent cell cycle reactivation through transcriptional regulation of Arabidopsis E2Fa by lateral organ boundary proteins[J].The plant cell,2011,23:3671-3683.
[6] 张昭阳.GWAS定位水稻成熟种子组培力相关基因及功能分析[D].武汉:华中农业大学,2018.ZHANG Z Y.Identification of genes related to rice matrue seed culturability by GWAS and functional analysis[D]. Wuhan:Huazhong Agricultural University,2018 (in Chinese with English abstract) .
[7] WANG L,XIE W,CHEN Y,et al. A dynamic gene expression atlas covering the entire life cycle of rice[J]. Plant journal for cell & molecular biology,2010,61:752-766.
[8] VINCENT S,VILHJ LMSSON B J,ALEXANDER P,et al. An efficient multi-locus mixed model approach for genome-wide association studies in structured populations[J]. Nature genetics,2012,44:825-830.
LMSSON B J,ALEXANDER P,et al. An efficient multi-locus mixed model approach for genome-wide association studies in structured populations[J]. Nature genetics,2012,44:825-830.
[9] 周非凡,刘瑜,常鑫磊,等.OsCPK12基因功能研究和互作蛋白筛选[J].华中农业大学学报,2019,38(6):48-55.ZHOU F F,LIU Y,CHANG X L,et al.Gene function of OsCPK12 and screening of its interaction proteins[J].Journal of Huazhong Agricultural University,2019,38(6):48-55(in Chinese with English abstract).
[10] BOLIBOK H,RAKOCZYTROJANOWSKA M. Genetic mapping of QTLs for tissue-culture response in plants[J].Euphytica,2006,149:73-83.
[11] BOTSTEIN D,RISCH N. Discovering genotypes underlying human phenotypes:past successes for mendelian disease,future approaches for complex disease[J]. Nature genetics,2003,33:228-237.
[12]TSUWAMOTO R,YOKOI S,TAKAHATA Y. Arabidopsis embryomaker encoding an AP2 domain transcription factor plays a key role in developmental change from vegetative to embryonic phase[J]. Plant molecular biology,2010,73:481-492.
[13] BOUYER D,ROUDIER F,HEESE M. Polycomb repressive complex 2 controls the embryo-to-seedling phase transition[J/OL]. PLoS genetics,2011,7:e1002014[2020- 12-08]. https://doi.org/10.1371/journal.pgen.1002014.
[14] BRATZEL F,L PEZ-TORREJ
PEZ-TORREJ N G,KOCH M. Keeping cell identity in Arabidopsis requires PRC1 RING-finger homologs that catalyze H2A monoubiquitination[J]. Current biology,2010,20:1853-1859.
N G,KOCH M. Keeping cell identity in Arabidopsis requires PRC1 RING-finger homologs that catalyze H2A monoubiquitination[J]. Current biology,2010,20:1853-1859.
[15] SUNKAR R,LI Y F,JAGADEESWARAN G. Functions of microRNAs in plant stress responses[J]. Trends in plant science,2012,17(4):196-203.
[16] SALEHIN M,BAGCHI R,ESTELLE M. SCFTIR1/AFB-based auxin perception:mechanism and role in plant growth and development[J]. Plant cell,2015,27(1):9-19.
[17] RINALDI M A,LIU J,ENDERS T A,et al. A gain-of-function mutation in IAA16 confers reduced responses to auxin and abscisic acid and impedes plant growth and fertility[J]. Plant molecular biology,2012,79:359-373.
[18] PLOENSE S E,WU M F,NAGPAL P,et al. A gain-of-function mutation in IAA18 alters Arabidopsis embryonic apical patterning[J]. Development,2009,136:1509-1517.
[19] CHANVIVATTANA Y,BISHOPP A,SCHUBERT D. Interaction of polycomb-group proteins controlling flowering in Arabidopsis[J]. Development,2004,131:5263-5276.
[20] CHEN C,LIU Q,ZHANG Y. Genome-wide discovery and analysis of microRNAs and other small RNAs from rice embryogenic callus[J]. RNA biology,2011,8:538-547.
[21] NEELAKANDAN A K,WANG K.Recent progress in the understanding of tissue culture-induced genome level changes in plants and potential applications[J].Plant cell reports,2012,31(4):597-620.
[22] MAJER C,HOCHHOLDINGER F.Defining the boundaries:structure and function of LOB domain proteins[J].Trends in plant science,2011,16(1):47-52.
[23] LIAN J,QIN Q,YU W,et al.Rice Dwarf virus P2 protein hijacks auxin signaling by directly targeting the rice OsIAA10 protein,enhancing viral infection and disease development[J/OL].PLoS Patho,2016,12:e1005847[2020-12-08].https://pubmed.ncbi.nlm.nih.gov/27606959/.DOI:10.1371/journal.ppat.1005847.
[24] VILLALOBOS L I,LEE S C,DE OLIVEIRA C.A combinatorial TIR1/AFB-Aux/IAA co-receptor system for differential sensing of auxin[J].Nature chemical biology,2012,8:477-485.
[25] NI J,WANG G H,ZHU Z X,et al.OsIAA23‐mediated auxin signaling defines postembryonic maintenance of QC in rice[J].Plant journal for cell & molecular biology,2011,68:433-442.
[26] IKEUCHI M,SUGIMOTO K,IWASE A.Plant callus:mechanisms of induction and repression[J].Plant cell,2013,25:3159-3173.
[27] HUANG X,ZHAO Y,WEI X,et al.Genome-wide association study of flowering time and grain yield traits in a worldwide collection of rice germplasm[J].Nature genetics,2012,44:32-39.
[28] WANG Q X,XIE W B,XING H K,et al.Genetic architecture of natural variation in rice chlorophyll content revealed by a genome-wide association study[J].Molecular plant,2015,8(6):946-957.
[29] CHAMBERLIN M,CUSHATT J.Morphogenic regulators baby boom and wuschel improve monocot transformation[J].Plant cell,2016,28(9):1998-2015.
[30] LI S J,YAN S,WANG A,et al.Identification of QTLs associated with tissue culture response through sequencing-based genotyping of RILs derived from 93-11 ×Nipponbare in rice (Oryza sativa)[J].Plant cell reports,2013,32(1):103-116.